�A���~�g�R���h���ADNA�̍\�����^�F�~�j�T�e���C�g�l�z��𗘗p�����~�g�R���h���A�}�[�J�[�̍�o
�P�D�͂��߂�
�@DNA����z�����̂����݂����������20���I�̂��b�ł���D�����̒��҂́C�e���T�C�~�g�R���h���A�Ɋ܂܂���}�X�^�[�T�[�N��DNA�̑S����z��Ƃ����v���W�F�N�g�Ɋւ���Ă����D�}�X�^�[�T�[�N��DNA�̓~�g�R���h���A�Q�m���̂قڑS�Ẳ���z�����ԗ�����̂ŁC����������e���T�C�~�g�R���h���A�Q�m���v���W�F�N�g�ƌĂ�ł����D�K���Ȃ��ƂɁC�}�X�^�[�T�[�N���S�̈�͊��ɃN���[�����i�������ă����_�t�@�[�W�x�N�^�[��R�X�~�h�x�N�^�[�ɑg�ݍ��݁C�K�v�ɉ����Ă�����咰�ۂ𗘗p���đ��B�����邱�Ƃ��\�j����Ă����D���Ƃ͊e�N���[���Ɋ܂܂��e���T�C�~�g�R���h���A�R����DNA����z�����ǂ��邾���ł���D�������C���̂���̌������ł͂���Ȃ�ɋZ�p�ƒm����v��������ł������̂ŁC��w�@���◯�w�������l���Q�����Ă����D������C��l�̊w���������̒S�����鉖��z������Ȃ�����Ă����D�����ɒ��҂��ʂ肩�������̂��C�ȉ��ɏq�ׂ錤���̔��[�ł������D
�Q�D�e���T�C�~�g�R���h���ADNA���̌J��Ԃ��z��
�@���̊w���́C32����ibp�j�̉���z�C�J��Ԃ������܂Ȃ������̈�����Ėʔ������Ă����̂ł���D���̌J��Ԃ��z��́C26S���{�\�[��RNA��`�q�ƃ��`�I�j��tRNA��`�q�ɋ��܂�Ă����D���̗̈�̉���z���}1�Ɏ����D
 |
�@�����̌������ł̓~�g�R���h���A�Q�m���\���̈Ⴂ��C���ꂪ�����Ȃ�`���Ƃ��Č����̂���Ɍ������Ă����̂ŁC���R�Ɓu�قȂ�~�g�R���h���A�^�ł����̌J��Ԃ��z��̈�̍\���͓������낤���C����Ƃ��Ⴄ���낤���v�Ƃ����c�_�ɂȂ����D
�@�j�J��Ԃ��z��́u�L�яk�݁v����
�@���̗̈�����ނ悤�ȃI���S�k�N���I�`�h�v���C�}�[��v���C�ʂȎ�������~�g�R���h���ADNA�^�C�v���قȂ邱�Ƃ����ɂ킩���Ă���e���T�C��߉��쐶���p����PCR���s�����D���̌��ʁC�傫���̈قȂ鑝��DNA�f�Ђ�����ꂽ�D�e�X��DNA�f�Ђ̉���z�����͂���ƁC�}1�Ŏ������n���ł�13��̌J��Ԃ��ł���i32 bp x 13��j���C���̑���2����8��܂ŁC�l�X�ȌJ��Ԃ��������������D���Ȃ킿�CPCR�Y���̒����̈Ⴂ�͌J��Ԃ����̈Ⴂ�ł��邱�Ƃ��킩�����i�}�Q�j�D
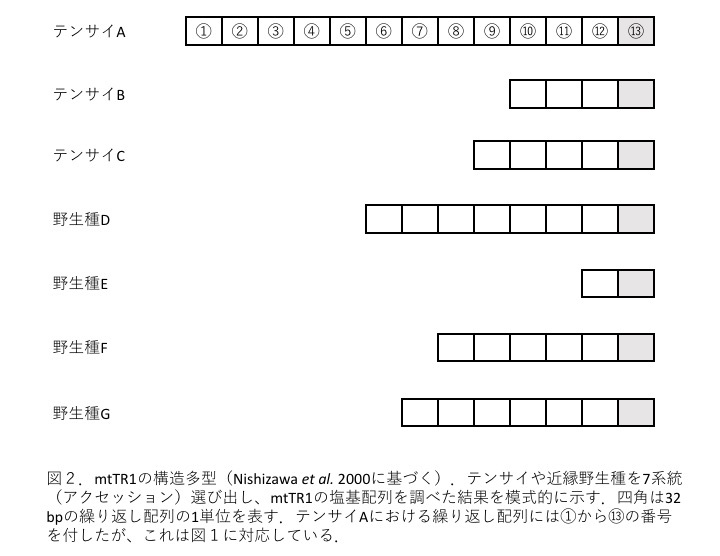 |
�@����ł́C�}�Q�Ŏ������悤�ȍ\�����^�̓~�g�R���h���A�^�ɌŗL�Ȃ̂��낤���D����Ƃ��C���s����ŁC�ɒ[�Ɍ����ΐ�����o�閈�ɕω�����̂��낤���D����ɂ��ẮC�����~�g�R���h���A�^�������Ƃ��킩���Ă���W�c�����C�S99�̂�����̍\���������Ƃ��m�F�����D�ǂ����C�����~�g�R���h���A�^�Ȃ炱�̗̈�������\���������悤�ł���D
�@�������������́C���̗̈悪������~�j�T�e���C�g�ł���i�����J��Ԃ��z��̒�����5 bp�ȉ��ł���}�C�N���T�e���C�g�ƌĂԂׂ��ł������낤[Buard and Vergnaud 1994]�j�C��������`�}�[�J�[�Ƃ��ė��p�ł��邱�Ƃ������Ă���D�ȍ~�C���̗̈��mtTR1�ƌĂԁD�o��y���q�g�̊jDNA���猩�����Ă���~�j�T�e���C�g�Ɠ��l�iHaber and Louis 1997�j�ɁCmtTR1�̌J��Ԃ��z��̖��[���i7 bp�j�͌J��Ԃ����n�܂钼�O�Ɠ�������z��ł���i�}�P�̎l�p�ň͂���z��j�D���̂��Ƃ���CmtTR1�̒a���ɂ́C�jDNA�̃~�j�T�e���C�g�a���Ɨގ��̋@�\���ւ���Ă���\���������D���łɓ����~�g�R���h���ADNA�Ń~�j�T�e���C�g�l�\�����m���Ă������ƁiLunt et al. 1998�j���킩�����Ƃ��͏������_�������C�A���~�g�R���h���ADNA�ł͏��߂Ă̔����ł������D
�A�j�~�j�T�e���C�g�͈ꃕ���ł͂Ȃ�
�@mtTR1�ȊO�Ƀ~�j�T�e���C�g�l�\�������邩�ǂ����C���肵���e���T�C�~�g�R���h���ADNA����z��������D���̌��ʁC����z�̂��͈̂قȂ邪�C30����66 bp�̔z�J��Ԃ������̈悪�V����3���������D�����ŁC���ꂼ���mtTR2����mtTR4�Ɩ��t���C�O���Ɠ��l�Ƀe���T�C��߉��쐶��̊Y���̈�������Ƃ���C��͂�J��Ԃ������قȂ邱�Ƃ��킩�����i�\�P�j
�\�P�@�e���T�C��߉��쐶��ɂ�����emtTR�̌J��Ԃ��z��
|
�n���^�A�N�Z�b�V�����̖��� |
mtTR���̂ƌJ��Ԃ��z��1�̒����ibp�j |
|||
|
mtTR1 (32) |
mtTR2 (33) |
mtTR3 (66) |
mtTR4 (30) |
|
|
�e���T�CA |
13 |
3 |
3 |
3 |
|
�e���T�CB |
4 |
3 |
2 |
4 |
|
�e���T�CC |
5 |
3 |
2 |
3 |
|
�쐶��D |
8 |
3 |
3 |
5 |
|
�쐶��E |
2 |
3 |
1 |
3 |
|
�쐶��F |
6 |
3 |
3 |
3 |
|
�쐶��G |
7 |
3 |
3 |
3 |
��Nishizawa et al. (2000)�Ɋ�Â��D
�\�P���CmtTR1�̌J��Ԃ�����2����13�܂ł�7�^�C�v����̂ɑ��C���̑���mtTR�ł͂�������3�^�C�v����������Ȃ����ƂɋC�����D����͉��ɋN������̂��낤���DmtTR1�Ƃ��̑���mtTR�̈Ⴂ�́C�~�g�R���h���ADNA��̈ʒu�ɂ���D���Ȃ킿�CmtTR1�݂̂�����ɑ����g�݊������Ă����Ǝv����DNA�̈����猩�����Ă���D���̂��Ƃ��s�������𑣐i���C��葽���̃^�C�v�ݏo���v���ƂȂ��Ă���̂�������Ȃ��D
�Q�D�e���T�C�ȊO�̐A���~�g�R���h���ADNA�Ƀ~�j�T�e���C�g�l�z��͑��݂��邩
�@�ȏ��_���Ƃ��ē��e�����iNishizawa et al. 2000�j���C�R�����̈�l����u�A���~�g�R���h���ADNA�ŁC�J��Ԃ��z��̐����ς���C�\�����^�������̂͏��߂Č����v�Ƃ������e�̃R�����g����������D���ʂ���C�O�̐A���~�g�R���h���ADNA�����҂ł��邱�Ƃ͖����������̂ŁC���������̌������F�߂Ă��炦���͔̂��ɂ��ꂵ�������D�������C����܂łȂ��N���~�j�T�e���C�g�ɋC�����Ȃ�������C���̗L���ׂ悤�Ƃ��Ȃ������̂��낤���D�l�X�ȐA���~�g�R���h���ADNA���r�����ꍇ�ɍۗ��\����̈Ⴂ�́C��`�q�̔z�u�̕ύX��C��r�ΏۂɂȂ��ŗL�̉���z��̑��݂ł���iGualberto and Newton 2017�j����C�܂��͂��������_�������҂ɒ��ڂ��ꂽ�D����ŁC�����͐A���~�g�R���h���ADNA����z����͖R�����C�~�j�T�e���C�g�����邩�ǂ����C���邢�͂��̑��^�ɂ��Ă͂قƂ�ǒ��ӂ��ނ��Ȃ������̂��낤�D�v����ɁC�N�����ׂ����Ƃ��Ȃ��Ǝv��ꂽ�D����ł́C�~�j�T�e���C�g�l�z��̑��݂ƍ\�����^�̓e���T�C�݂̂̓���Ȏ���Ȃ̂��낤���C����Ƃ��A���~�g�R���h���ADNA�ɕ��ՂȂ̂��낤���D
�@�A���~�g�R���h���ADNA�W�̘_����ǂ�ł���ƁC���̐A����ɂ��~�g�R���h���A�~�j�T�e���C�g�����݂��C�������\�����^������\���ɋC�������D�Ⴆ�Corf138��orf125�Ƃ����~�g�R���h���A��`�q������iBonhomme et al. 1992; Iwabuchi et al. 1999�j�D�����͓��{�̃_�C�R������C�������͓��{�̃_�C�R����e�Ƃ���i�^�l���猩��������`�q�ŁC����z�݂��ɂƂĂ��悭���Ă���D���҂̉���z���̈Ⴂ��orf125�ɂ�����39 bp�̌����ł��邪�C���ꂪ�~�j�T�e���C�g�̂悤�ɂ݂���̂ł���D���Ȃ킿�Corf138�ł͂���39 bp�z��Ƃ悭�����z��3��J��Ԃ���Ă���̂ɑ��Corf125�ł�2��̌J��Ԃ��ŁC���x1���J��Ԃ����������Ă���D���������؋�����C�~�j�T�e���C�g�l�̈悪���ɂ����邾�낤�Ƃ������G���D
�@�}�X�^�[�T�[�N��DNA�̑S����z��ǂ��ꂽ�A���킪��������������v�炢�C�e���T�C�ȊO�̐A���킩��~�g�R���h���A�~�j�T�e���C�g��T�����Ƃɂ����D���̌��ʁC���ׂ�10�̐A����S�Ă��~�j�T�e���C�g�l�̍\�������������iHonma et al. 2011�j�D�����āC���������~�j�T�e���C�g�ɑ��^�����邩�ǂ����������D�����ł́C�A�u���i���̌��ʂɂ��ďЉ��D
�@�A�u���i���A���̈�`�I�W�́C�������Z�̎O�p�`�ŕ\�����Ƃ��ł���i�}3�j�D
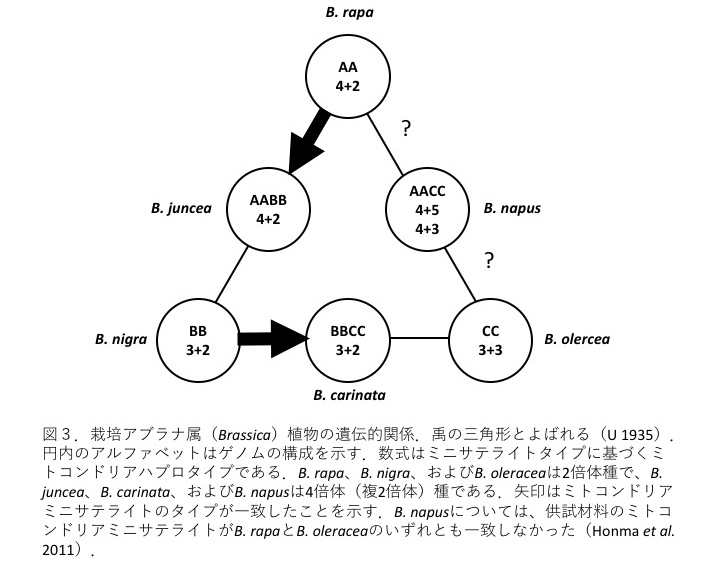 |
�Z�C���E�i�^�l�̃~�g�R���h���A�}�X�^�[�T�[�N��DNA�ォ��6�̃~�j�T�e���C�g�l�̈悪���������̂ŁC�}�R�Ɏ���6�̐A����̊Y���̈�ׂĂ݂��D�c�O�Ȃ���C���ׂ�6�̃~�j�T�e���C�g�̂����C4�͑S�Ă̋����ޗ��œ���̌J��Ԃ����ł������D����C�c���2�ɂ��Ă�2����3�^�C�v�����������D�~�g�R���h���A�̕ꐫ��`�܂���ƁC���e�ԂŃ~�g�R���h���ADNA�̈�`�I�g�݊����͂Ȃ��D�����ł���Ȃ�C���^����������2�̃~�j�T�e���C�g�̈�͊��S�A���ƂȂ�D�]���āC�������܂Ƃ߂ăn�v���^�C�v�Ƃ��Ĉ����Ă��ǂ����ƂɂȂ�D�Ⴆ�C����ޗ��ɂ����Ĉ�̃~�j�T�e���C�g�̈�ɂ�����J��Ԃ��z��̐���4�ŁC������̗̈悪2�ł���C�����4+2�Ƃ����n�v���^�C�v�Ƃ݂Ȃ��Ă悢�DB. rapa�͋�������6�A�N�Z�b�V�������S�Ă�4+2�ł������iHonma et al. 2011�j�D����CB. nigra��3+2�ŁCB. juncea��4+2�ł������D���Ȃ킿�CB. juncea�̃~�g�R���h���ADNA�͂ǂ��炩�Ƃ�����B. rapa�Ɏ��Ă��邱�ƂɂȂ�i��`�I�g�݊������Ȃ��Ƃ���CB. juncea�̓�̃~�g�R���h���A�~�j�T�e���C�g�̕Е���B. rapa�R���ł������B. nigra�R���Ƃ������߂͐������Ȃ��j�D���l�ɁCB. carinata��3+2�ŁCB. oleracea��3+3�ł���CB. carinata�̃~�g�R���h���ADNA��B. nigra�Ɏ��Ă���D�ȏ�̌��ʂ́C�t�Α�DNA�̉�͌��ʁiErickson et al. 1983; Palmer et al. 1983�j�Ɨǂ���v���Ă����D���Ȃ킿�CB. juncea��B. carinata����������ߒ��ɂ����Ď�q�e�i��e�j�ƂȂ����̂́C���ꂼ��B. rapa��B. nigra�ł������Ƃ���ʐ����x�����錋�ʂƂȂ����D����������̂́CB. napus���猩������4+5��4+3�Ȃ�n�v���^�C�v�ł���D�����͋����ޗ��̒����猩���炸�C�]����B. napus�̃~�g�R���h���A��������̑c��Q�{�̎�ɗR������̂��͂킩��Ȃ������DB. napus�̎�q�e���͑�ϓ���悤�ŁiSong et al. 1988; Flannery et al. 2006; Allender and King 2010�j�C����ȏ�̓A�u���i���̐��ƂɔC���悤�ƂȂ����D
�R�D�~�g�R���h���A�~�j�T�e���C�g�̗��p
�@����܂łɗl�X�ȐA����ɂ����ă~�g�R���h���A�}�[�J�[���J������Ă���D��r�I�������̂Ƃ��āC����~�g�R���h���A�^�ɌŗL�̓�����W�I�Ƃ�����̂�����D�Ⴆ�CA�Ƃ����~�g�R���h���A�^�ɌŗL��DNA����z���W�I�Ƃ���}�[�J�[��p����ƁC�uA�ł���v�������́uA�łȂ��v�Ƃ����������D�������CA�łȂ��Ȃ�B���CC���C���邢��D���C�Ƃ����悤�ȏڍׂ�m�肽���ꍇ�́C���̃}�[�J�[�ł͕s�\���ł���D����ɑ��C�~�j�T�e���C�g�͌J��Ԃ����̈Ⴂ�Ɋ�Â��Ă���̂ŁC�����̃~�g�R���h���A�^�ɑΉ��ł���V�X�e���ł���D���p�I�ɂ��C�~�j�T�e���C�g�͌J��Ԃ��z��1����r�I�����i30����66 bp�j���߁C�Q���d�C�j�����̈Ⴂ���킩��₷���Ƃ������_������D���̂��߁C��y�Ƀ~�g�R���h���A�^��m�邽�߂̎w�W�Ƃ��ĕ֗��ł���D�Ⴆ�C�e���T�C�i����ǂ̌���Ń~�g�R���h���A�^�ʂ��邽�߂ɗ��p����Ă���iCheng et al. 2009�Ȃǁj�D�ł��͂�����̂��C�����̃~�g�R���h���A�^�������܂�鑽���̎����������悤�ȏW�c��`�w��i���w�I�Ȍ�����ʂł���D�e���T�C�C�K�[�f���r�[�g�C���邢�̓t�_���\�E�ƁC�߉��쐶��̈�`�I�W���~�g�R���h���A�^�̊ϓ_���璲�ׂ��Ă���iNishizawa et al. 2007; Cheng et al. 2011; Yoshida et al. 2012�j�D�t�����X�Ɏ�������߉��쐶����ޗ��Ƃ������ԁE�i����`�w�I�����ɂ����p����Ă���iFievet et al. 2007; Fenart et al. 2008; Arnaud et al. 2009; De Cauwer et al. 2010; De Cauwer et al. 2012�j�D�e���T�C��߉��쐶������킹���O���[�v����́C4�̃~�j�T�e���C�g�̃n�v���^�C�v�ɂ��20��ޒ��x�̃~�g�R���h���A�^���������ꂽ�D���̃}�[�J�[�͍����g���Ă���D
�@
�S�D���Ƃ���
�@����z���͋Z�p�͓��i�����ł���C�����ɑ����̌��̂̑S����z������悤�Ȏ���ɂȂ�C��`�q�����������I�яo���Ĕ�r����Ƃ��������͏��ł��邩������Ȃ��D�����Ȃ�}�[�J�[�Ƃ��Ẵ~�g�R���h���A�~�j�T�e���C�g�̉��l�͂قƂ�ǂȂ��Ȃ邾�낤�i�~�g�R���h���ADNA���\���ψق���d�g�ׂ݂郂�f���Ƃ��Ă͗L�p�ł͂Ȃ����ƍl����j�D
�@����ŁC�{�e�Ɍ����G�s�\�[�h�Ƃ��Ă̖ʔ����𑽏������Ă���������Ȃ�C�L�^�Ƃ��Ďc���Ӌ`�����낤�D���ɔ��[���Ӑ}�I�ł͂Ȃ��������Ƃ������������D���҂�́C�~�j�T�e���C�g��T���ړI�Ńe���T�C�~�g�R���h���ADNA�̉���z��ׂĂ����킯�ł͂Ȃ��D�ނ���C�S�������w������X�J��Ԃ��z��ɋC�����i�����\�͂Ƌ��R�j�C������u�ʔ����v�Ɗ��������Ɓi���E�Z���X�j���_�@�ƂȂ����D������w�i�Ƃ��ďd�v�������̂́C�����O���[�v�S�̂Ń~�g�R���h���ADNA�\�����^�ɋ���������C�~�j�T�e���C�g�ɂ�����J��Ԃ����̈Ⴂ�����̎w�W�ɂȂ�\����S���������ɗ����ł������Ƃ���������i���_�̋��L�j�D�Ⴆ�C������PI���u�܂�Ȃ��̂ŁC����Ȏ����͂�߂ăv���W�F�N�g�ɏW�����Ȃ����v�Ɩ����Ă�����i���ۂ͋t�ŁC�f�[�^�����Ėʔ������Ă��ꂽ�j�C�����͑������C��b�����ɂ����p�ɂ��g����~�g�R���h���A�}�[�J�[�͒a�����Ȃ��������낤�D�����Ƃ��C���̂悤�ȋ��R�̔������E���グ��]�T�̂��������Ƃ���Ԃ̍K�^�������̂�������Ȃ��D
�@���ȏ��ɍڂ�悤�Ȕ����E�����ɂ������R�̗v�f�������͉̂Ȋw�j�̓`����Ƃ���ŁC�w�����N�̒��ɂ͂��������X�g�[���[�ɓ���Č������u���҂�����Ǝv���D�{�e�ŏЉ�������́u���l�^�v�ɉ߂��Ȃ����C�V���������̎�q���g�߂ɂ��邱�Ƃ��`���K���ł���D
References
Allender, C.J. and G. J. King, 2010 Origins of the amphiploid species Brassica napus L. investigated by chloroplast and nuclear molecular markers. BMC Plant Biol. 10, 54.
Arnaud, J.F., S. Fenart, C. Gode, S. Deledicque, P. Touzet and J. Cuguen, 2009 Fine-scale geographical structure of genetic diversity in inland wild beet populations. Mol. Ecol. 18, 3201-3215.
Bonhomme S., F. Budar, D. Lancellin, I. Small and G. Pelletier, 1992 Sequence and transcript analysis of the Nco 2.5 ogura-specific fragment correlated with cytoplasmic male sterility in Brassica cybrid. Mol Gen Genet 235, 340–348
Buard J. and G. Vergnaud, 1994 Complex recombination events at the hypermutable minisatellite CEB1 (D2S90). EMBO J. 13, 3203-3210.
Cheng, D., K. Kitazaki, D. Xu, T. Mikami and T. Kubo, 2009 The distribution of normal and male-sterile cytoplasms in Chinese sugar-beet germplasm. Euphytica 165, 345-351.
Cheng, D., Y. Yoshida, K. Kitazaki, S. Negoro, H. Takahashi, D. Xu, T. Mikami and T. Kubo, 2011 Mitochondrial genome diversity in Beta vulgaris L. ssp. vulgaris (Leaf and Garden Beet Groups) and its implications concerning the dissemination of the crop. Genet. Res. Crop Evol. 58, 553-560.
De Cauwer, I., M. Dufay, J. Cuguen and J.F. Arnaud, 2010 Effects of fine-scale genetic structure on male mating success in gynodioecious Beta vulgaris ssp maritima. Mol. Ecol. 19, 1540-1558.
De Cauwer, I., M. Dufay, B. Hornoy, A. Courseaux and J.F. Arnaud, 2012 Gynodioecy in structured populations: understanding fine-scale sex ratio variation in Beta vulgaris ssp maritima. Mol. Ecol. 21, 834-850.
Erickson, L. R., N.A. Straus and W. D. Beversdorf, 1983 Restriction patterns reveal origins of chloroplast genomes in Brassica amphiploids. Theor. Appl. Genet. 65, 201–206.
Fenart, S., J.F. Arnaud, I. de Cauwer and J. Cuguen, 2008 Nuclear and cytoplasmic genetic diversity in weed beet and sugar beet accessions compared to wild relatives: new insights into the genetic relationships within the Beta vulgaris complex species. Theor. Appl. Genet. 116, 1063-1077.
Fievet, V., P. Touzet, J.F. Arnaud and J. Cuguen, 2007 Spatial analysis of nuclear and cytoplasmic DNA diversity in wild sea beet (Beta vulgaris ssp maritima) populations: do marine currents shape the genetic structure? Mol. Ecol. 16, 1847-1864.
Flannery, M. L., F. J. G. Mitchell, S. Coyne, T. A. Kavanagh, J. I. Burke, N. Salamin, P. Dowding, T. and R. Hodkinson, 2006 Plastid genome characterisation in Brassica and Brassicaceae using a new set of nine SSRs. Theor. Appl. Genet. 113, 1221–1231.
Gualberto J.M. and K.J. Newton, 2017 Plant mitochondrial genomes:dynamics and mechanisms of mutation. Annu. Rev. Plant Biol. 68, 225-252.
Haber, J.E. and E.J. Louis, 1998 Minisatellite origins in yeast and humans. Genomics 48, 132-135.
Honma, Y., Y. Yoshida, T. Terachi, K. Toriyama, T. Mikami and T. Kubo, 2011 Polymorphic minisatellites in the mitochondrial DNAs of Oryza and Brassica. Curr. Genet. 57, 261-270.
Iwabuchi, M., N. Koizuka, H. Fujimoto, T. Sakai and J. Imamura, 1999 Identification and expression of the kosena radish (Raphanus sativus cv. Kosena) homologue of the ogura radish CMS-associated gene, orf138. Plant Mol. Biol. 39, 183-188.
Lunt, D. H., L. E. Whipple and B. C. Hyman, 1998 Mitochondrial DNA variable number tandem repeats (VNTRs): utility and problems in molecular ecology, Mol. Ecol. 7, 1441-1455.
Nishizawa, S., T. Kubo and T. Mikami, 2000 Variable number of tandem repeat loci in the mitochondrial genomes of beets. Curr. Genet. 37, 34-38.
Nishizawa, S., T. Mikami and T. Kubo, 2007 Mitochondrial DNA phylogeny of cultivated and wild beets: Relationships among cytoplasmic male-sterility-inducing and nonsterilizing cytoplasms. Genetics 177, 1703-1712.
Palmer, J. D., C. R. Shields, D. B. Cohen and T. J. Orton, 1983 Chloroplast DNA evolution and the origin of amphidiploid Brassica species. Theor. Appl. Genet. 65, 181–189.
Song, K. M., T. C. Osborn and P. H. Williams, 1988 Brassica taxonomy based on nuclear restriction fragment length polymorphisms (RFLPS) 1 Genome evolution of diploid and amphidiploid species. Theor. Appl. Genet. 75, 784–794.
U, N., 1935 Genome analysis in Brassica with special reference to the experimental formation of B. napus and peculiar mode of fertilisation. Jpn. J. Botany 7, 389–452.
Yoshida Y, M. Matsunaga, D. Cheng, D. Xu, Y. Honma, T. Mikami and T. Kubo 2012 Mitochondrial minisatellite polymorphisms in fodder and sugar beets reveal genetic bottlenecks associated with domestication. Biol. Plant. 56, 369-372.
�i2019.6.5�j