植物ミトコンドリアゲノムの姿
はじめに
「ゲノム」の定義は,時代により,人により異なるが,ある生物の持つ遺伝情報1セットとして間違いなかろう.そうすると,ミトコンドリアは(細胞内共生以前は独立した生物だったとはいえ,今では)生物ではなく細胞小器官であるから,ミトコンドリアゲノムとはおかしな言葉であるが,研究者はこれをミトコンドリアに含まれる遺伝情報1セットという意味で使っている.かつて,細胞質遺伝学の研究者はミトコンドリアが保持する遺伝情報のことをchondriomeと称していた.しかしこの言葉は廃れてしまい,今ではミトコンドリアゲノムという言葉がそれに取って代わっている.
ここでいう遺伝情報とは,タンパク質コード領域のみならず,遺伝子間領域なども全て含むDNA塩基配列情報のことである.したがって,ミトコンドリアゲノムとはミトコンドリアDNA(mtDNA)塩基配列情報のことを指す.それゆえ,ミトコンドリアゲノムのことを知りたければmtDNAを調べねばならない.
mtDNAの姿は生物種毎に大きく異なる.しかしながら,広く紹介されているのはヒトやマウスのmtDNAばかりであり,その他の生物種については目にする機会が少ない.生物界を広く見渡した場合,ヒトmtDNAの特徴がそのまま他生物種に適用できるとは限らないので,mtDNAに関連する研究を行う場合には多様性に注意する必要がある.
被子植物mtDNAに関しては未解決の問題が多く残されており,その姿を一言で説明するのは大変難しい.そのため,専門書や学術論文であっても大まかな説明しかされていないのが普通である.ここでは,被子植物mtDNAについて講義内容を復習・補足する.最小限の事例の紹介であったり,出典が総説であるので,興味があれば文献検索により原典を調べることをおすすめする.なお,被子植物から系統発生学的に離れるに従って事情が異なってくることに注意されたい.
植物mtDNAの姿:初期の研究
古い総説1によれば,当初は被子植物mtDNA分子もヒトのように均一だと思われていたようである.しかしながら,複数のDNA分子種が混在していることが,研究が進むようになって指摘されるようになった.
分析の一例として,電子顕微鏡による観察がある.テンサイmtDNAでは,分子量の異なる環状分子と線状分子が観察された2, 3.同条件で調製された色素体DNAはほぼ均一な環状分子であったというから4,テンサイmtDNAはサイズや形状の異なる不均一な分子の集合体であるとみてよい.観察された環状DNA分子は,高分子量DNA(概ね3.0 kbp以上)とそれに満たない低分子量のプラスミド様DNAに分けられた.高分子量DNA群とプラスミド様DNA群のいずれにもいくつかのサイズの異なるピークが見られるので,各々のクラスにはさらに複数種のDNA分子が含まれると考えられた.
別な分析例として,mtDNA制限酵素消化物のアガロースゲル電気泳動像観察がある.分画されたDNA断片のパターンは非常に複雑で,分子量の近い隣り合うバンドの濃淡がしばしば極端に異なっていた2.これは,反復配列が存在するか,複数のDNA分子種が試料に混在する際に見られるパターンによく似ており,電子顕微鏡観察の結果と矛盾しない.
植物mtDNAの姿:マスターサークルモデル
1984年にカブ(Brassica campestris)を使った以下のような実験データが発表された5.まず,mtDNAを制限酵素で消化し,生じた断片を,それぞれプラスミドベクターにクローン化した.次に,これらをプローブにしてmtDNAもしくは他クローンに対するサザンハイブリダイゼーションを行って,物理構造を少しずつ明らかにしていった.用いるプローブを変えながらこれを繰り返すと,最終的に全体が218 kbpの一つの環状構造としてまとまった.この環状構造体をマスターサークル(マスター染色体)(MC)という.MCはミトコンドリアゲノムの遺伝情報をほぼ全て網羅していると説明されている.
カブMC上には,2 kbpの反復配列2コピーが直列方向に配置されている.この反復配列間で相同組み換えが生じていることが示されており,その結果218 kbpのMCは135 kbpと83 kbpの二つの環状分子に分割されうるという.被子植物ミトコンドリアゲノム構造に対するこのような説明をMCモデルと呼ぶことにする.
同様な実験手法を用いると,他植物種においてもMCを構築できる.例えば,テンサイではmtDNAを4塩基認識の制限酵素で部分分解して,いくつかのクローン同士が相互に重なるようなDNAライブラリーを作成し,染色体歩行によりMCを描き出すことに成功した6.近年の(いわゆる次世代シーケンス解析に代表される)High-throughput解析による植物ミトコンドリアmtDNA塩基配列決定も,原理的には同じである.
マスターサークルモデルの問題点
多くの教科書では,植物ミトコンドリアゲノムをMCモデルで説明している.一方,MCモデルには以下のような問題点もある.
マスターサークル=ミトコンドリアゲノム?
実験手法からわかるように,MCはmtDNA断片をつないでいくと机上に現れる環状構造である.ここにはほぼ全ての塩基配列が反映されている.「ほぼ」,というのはMCに含まれないmtDNA断片が存在するからである.例えば,低分子量のプラスミド様DNAは,MCと相同性を持たないことが多いので,MCには含まれない.次のような例を見ると,このようなDNA分子もミトコンドリアゲノムに含めた方が良いと感じるだろう.すなわち,トウモロコシ2.3 kbp線状プラスミド様DNAはトリプトファンtRNA遺伝子をコードするが,これはトウモロコシMC上に存在しない遺伝子である7.よってMCだけで遺伝子構成を議論すると,「トウモロコシミトコンドリアにはトリプトファンtRNA遺伝子が存在しない」という誤った結論を導く.同様な例は今のところ知られていないので,これは特殊なケースかもしれない.しかしながら,MCはmtDNAの(大部分ではあるけれども)一部分に過ぎないことは憶えておいた方が良いと思う.
マスターサークルが構築できない
まずはMC構築過程を簡単に紹介するため,染色体歩行によりミトコンドリアDNA解析を行った例を図1に示す.mtDNAの染色体歩行を行う場合,既知のミトコンドリア遺伝子を起点とすることが多い.図1では,矢印を起点として隣接するDNAクローンを次々に結合させ,最後に起点に戻った.この結果は,単一の環状mtDNA分子上を歩行したと解釈できる.このようなシンプルなmtDNAを持つと報告された植物種もある8など.
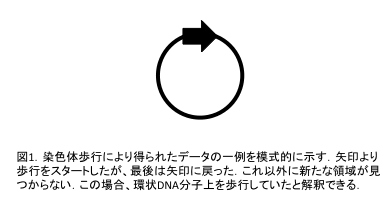
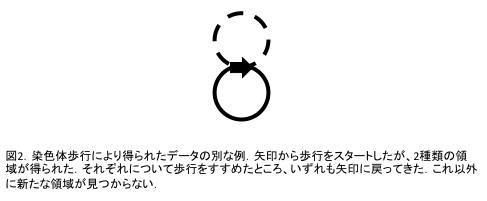
図2の例は,反復配列間での相同組み換えを考慮しなくてはならないものである.ここでは,起点である矢印から,2種類の塩基配列(実線と破線)が続いている.この場合,それぞれについて,2方向に歩行を進める.その結果,いずれも起点に戻ってきた.
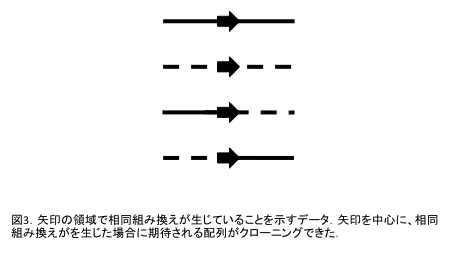
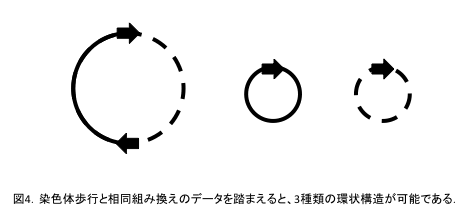
矢印を含むクローンの解析から,図3のような4種類の構造が明らかになった.すなわち,実線,破線に加え,矢印を境に破線と実線が入れ替わったような合計4種類である.このような4種類の構造が生ずるには,矢印を介した相同組み換えを仮定するのがよい.図2と図3より,得られたデータは図4のように解釈される.すなわち,図2を(矢印は2回通るが)一筆書きすると,実線と破線を含む矢印を2つ持つ大きな環状構造ができる.実線,もしくは破線だけでも,それぞれ矢印を1つ持つ小環状構造ができる.これより,染色体歩行に供したmtDNA試料は,これら3種類のDNA分子の混合物であったと解釈し,最も大きな環状構造をMCと呼ぶ.植物種によっては,矢印が3コピー以上のもの,あるいは矢印が数種類あるものが報告されている9など.その結果として,mtDNAを構成するDNA分子種が増え,非常に複雑になる.
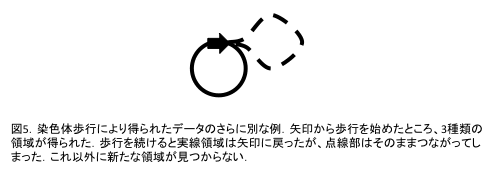
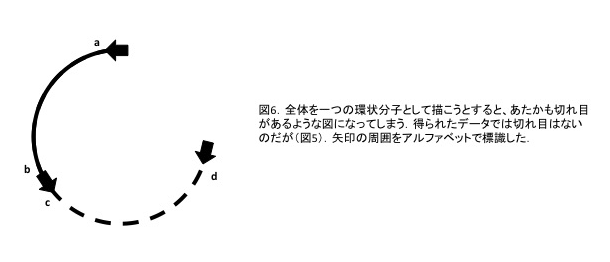
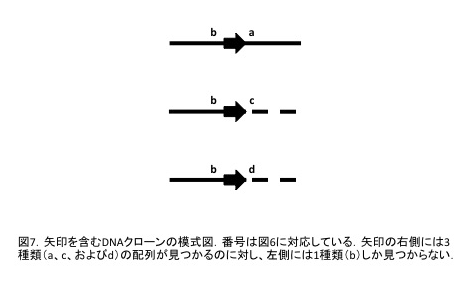
以上の例はMCが構築できる例である.一方,クローニングには何ら支障が無いにも関わらず,MCが構築できない例がある.そのような染色体歩行の例を図5に示す.図2にならい一筆書きを試みても,実線と破線の両方を含む環状構造は構築できない.さらに言えば,破線部を含むいかなる環状構造も構築不可能なのである.図5を書き改めたものが,図6である.矢印の途切れたところからこれ以上歩行を続けても領域b-aが続けて現れるだけなので,省略してある.図7に,矢印を含む領域を抜き書きした.実際にこのような例がニンジンで報告された10.
別な例として,環状分子は複数できるが,一つのMCは構築できないという報告がある.これは,相同組み換えを生ずるような共通配列が環状分子間に見つからないことによる.キュウリでは,解析結果が3つの環状分子としてまとめられ,それらが一つの大きな環状分子になることはないという11.
マスターサークルは実在するか?
実は,植物ミトコンドリア研究者のほとんどはMCが植物細胞(少なくとも栄養器官)で大量に存在するとは考えていない.電子顕微鏡観察により発見された高分子量DNA分子には,MC大のものが含まれていたが,その頻度は低いし,これがMCであるという証拠もない.MC大のDNA分子が見つからないという報告もある12, 13.細胞によっては,個々のミトコンドリア内におけるDNA量は遺伝子1セット分(すなわちMC)に満たないという14, 15.MCをゲルで分画(パルスフィールド電気泳動)しようと試みても,そのようなDNA分子は認められない12など.よって,MCが実在することを支持するデータは今のところ見当たらないことになっている.
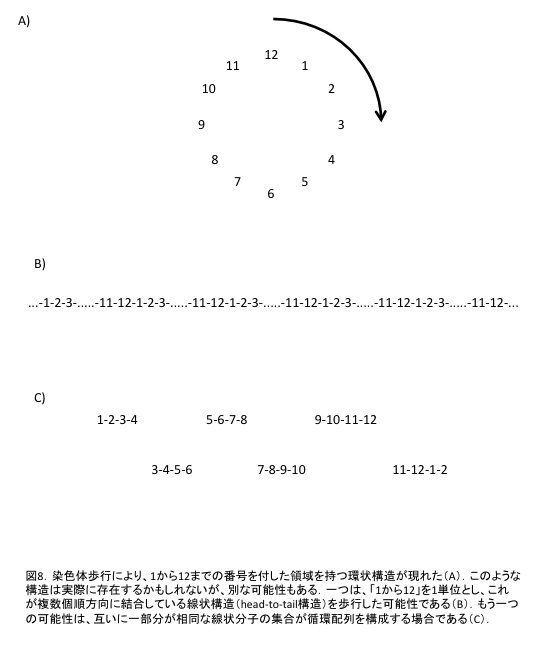
採用する実験手法によって結果が異なるのはなぜだろうか.総説12における議論によれば,染色体歩行により環状構造が現れるのは,実際のDNA分子が環状である以外に,MC大の線状DNA分子がhead-to-tailでコンカテマーを形成したり,線状DNA分子の集合体が(MCに相当する情報量を持つ)循環配列を構成する場合がある(図8).さらに、細胞の状態によってmtDNA分子の姿や量は大きく変化する16, 17, 18など.MCが存在するとしても,それは植物mtDNA構造の一側面に過ぎないのかもしれない.
マスターサークルの使い方
以上のようにMCは植物mtDNAの実際の姿を反映していない可能性が極めて高い.一方でMCが構築できたということは,その供試材料のmtDNAの大部分がクローン化できて,ミトコンドリア遺伝子構成の全容がおおよそ明らかにできたということを意味する.すなわち,任意に選び出したmtDNA断片は,ほぼ間違いなくMC上のどこかに対応するのである.よってMCは植物ミトコンドリアゲノムの優れた表現方法といえよう.研究目的によっては,ミトコンドリア遺伝子構成が主眼であって,実際のmtDNAの姿を追求する必要は無い.例えば,系統間や植物種間のミトコンドリア遺伝子構成についての議論であれば,MCで十分であろう.
引用文献
1. Leaver CJ, Gray MW, Mitochondrial genome organization and expression in higher plants, Annual Review of Plant Physiology, 33: 373-402, 1982.
2. Mikami T, Harada T, Kinoshita T, Heterogeneity of circular mitochondrial DNA molecules from sugar beet with normal and male sterile cytoplasms, Current Genetics, 10: 695-700, 1986.
3. Dudareva NA, Kiseleva EV, Boyarintseva AE, Maystrenko AG, Khristolyubova NB, et al., Structure of the mitochondrial genome of Beta vulgaris L., Theoretical and Applied Genetics, 76: 753-759, 1988.[電子顕微鏡観察の条件について,問題点が指摘されている12]
4. 三上哲夫,雄性不稔植物のミトコンドリアゲノムにみられる構造変異,化学と生物,23: 514-517, 1985.
5. Palmer JD, Shields CR, Tripartite structure of the Brassica campestris mitochondrial genome, Nature, 307: 437-440, 1984.
6. Kubo T, Satoh Y, Muro T, Kinoshita T, Mikami T, Physical and gene organization of mitochondrial DNA from the fertile cytoplasm of sugarbeet (Beta vulgaris L.), Current Genetics, 28: 235-241, 1995.
7. Leon P, Walbot V, Bedinger P, Molecular analysis of the linear 2.3 kb plasmid of maize mitochondria: apparent capture of tRNA genes, Nucleic Acids Research, 17: 4089-4099, 1989.
8. Palmer JD, Herbon LA, Unicircular structure of the Brassica hirta mitochondrial genome, Current Genetics, 11: 565-570, 1987.[ただし染色体歩行は行っていない]
9. Lonsdale DM, Hodge TP, Fauron CMR, The physical map and organization of the mitochondrial genome from the fertile cytoplasm of maize, Nucleic Acids Research, 12: 9249-9261, 1984.
10. Robison MM, Wolyn DJ, Complex organization of the mitochondrial genome of petaloid CMS carrot, Molecular Genetics and Genomics, 268: 232-239, 2002.
11. Alverson AJ, Rice DW, Dickinson S, Barry K, Palmer JD, Origins and recombination of the bacterial-sized multichromosomal mitochondrial genome of cucumber, The Plant Cell, 23: 2499-2513, 2011.
12. Bendich AJ, Reaching for the ring: the study of mitochondrial genome structure, Current Genetics, 24: 279-290, 1993.
13. Backert S, Nielsen BL, Börner T, The mystery of the rings: structure and replication of mitochondrial genomes from higher plants, Trends in Plant Science, 2: 477-483, 1997.
14. Satoh M, Nemoto Y, Kawano S, Nagata T, Hirokawa H, Kuroiwa T, Organization of heterogeneous mitochondrial DNA molecules in mitochondrial nuclei of cultured tobacco cells, Protoplasma, 175: 112-120, 1993.
15. Preuten T, Cincu E, Fuchs J, Zoschke R, Liere K, Börner T, Fewer genes than organelles: extremely low and variable gene copy numbers in mitochondria of somatic plant cells, The Plant Journal, 64: 948-959, 2010.
16. Cheng N, Lo Y-S, Ansari MI, Ho KC, Jeng ST, Lin NS, Dai H, Correlation between mtDNA complexity and mtDNA replication mode in developing cotyledon mitochondria during mung bean seed germination, New Phytologist, 213: 751-763, 2016.
17. Backert S, Börner T, Phage T4-like intermediates of DNA replication and recombination in the mitochondria of the higher plant Chenopodium album (L.), Current Genetics, 37: 304-314, 2000.
18. Kuroiwa T, Fujie M, Kuroiwa H, Studies on the behavior of mitochondrial DNA. Synthesis of mitochondrial DNA occurs actively in a specific region just above the quiscent center in the root meristem of Pelargonium zonale, Journal of Cell Science, 101: 483-493, 1992.
(2017.3.14)